吉林免疫熒光全景掃描
在土壤生物學研究中,全景掃描技術 實現了對土壤生態系統的多尺度、高精度可視化分析。通過X射線微斷層掃描(Micro-CT) 結合熒光原位雜交(FISH)技術,研究者能夠三維重構土壤剖面,精確解析土壤團聚體結構、孔隙網絡連通性以及微生物的空間分布模式。例如,在農田土壤研究中,全景掃描揭示了大孔隙(>50μm) 對作物根系延伸的關鍵作用,而微孔隙(<10μm)則***影響水分保持與養分擴散。同時,微生物群落的空間異質性分布 被發現與有機質分解效率直接相關——放線菌和***菌絲傾向于定殖于有機質富集的孔隙邊緣,驅動碳氮循環。
對極地苔原植被全景掃描,評估氣候變暖對其覆蓋度的影響。吉林免疫熒光全景掃描
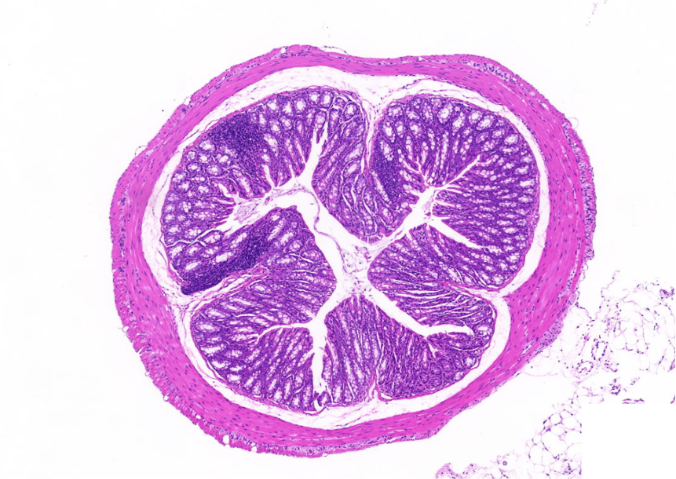
0. 病毒生態學研究中,全景掃描技術用于調查病毒在不同生態環境中的分布與傳播路徑,通過采集水體、空氣、動植物樣本進行全景掃描,識別病毒的種類、數量及宿主范圍。結合宏基因組學分析,揭示病毒與宿主及其他微生物的相互作用,例如在研究海洋病毒時,全景掃描發現了病毒在海洋浮游生物中的***分布及對浮游生物群落結構的調控作用,為理解海洋生態系統的物質循環和能量流動提供了新視角,也為防控病毒性傳染病的暴發提供了預警依據。吉林熒光三標全景掃描市場價格全景掃描追蹤胚胎著床,觀察胚泡與子宮內膜的識別及附著過程。

在神經再生研究中,全景掃描技術通過多模態動態成像系統實現了對神經修復過程的高精度時空解析。該技術整合雙光子***顯微術(2P-LSM)、光片熒光顯微鏡(LSFM)和擴散張量磁共振成像(DTI),可在單細胞水平追蹤神經干細胞***→軸突定向生長→突觸重建的全鏈條過程。以脊髓損傷模型為例,轉基因熒光標記的全景掃描顯示:①NT-3神經營養因子能誘導損傷區室管膜細胞轉分化(DCX+/Nestin+),24小時內形成再生微環境;②再生軸突以"跳躍式生長"模式(平均速度1.2μm/h)穿越膠質瘢痕,其生長錐的絲狀偽足動態變化(每秒3次伸縮)可通過超分辨成像(STED)清晰捕捉。結合行為學-電生理同步分析發現,當再生軸突與遠端V2a中間神經元形成功能性突觸(突觸素SYN1熒光強度>800AU)時,后肢運動功能(BBB評分)可恢復至8分以上。這些數據指導了"生物支架-生長因子"協同策略的優化:含層粘連蛋白通道的3D打印支架使軸突再生效率提升4倍。***突破是采用石墨烯量子點標記的全景掃描,***在***觀察到線粒體轉運對軸突再生的能量供應機制(損傷后線粒體沿微管向生長錐聚集速度加快50%)。
在植物逆境生理學研究中,全景掃描技術 通過多維度表型組-生理組聯合分析,系統揭示了植物應對環境脅迫的適應性策略。該技術整合 高光譜成像(400-2500nm)、激光共聚焦顯微術 和 X射線斷層掃描,實現了從***到細胞水平的動態響應監測。以小麥抗旱研究為例,根系原位全景掃描 顯示:在土壤含水量降至12%時,抗旱品種能快速啟動 "深根系化" 策略(主根伸長速率提高3倍),并通過 根冠黏液層增厚(掃描電鏡顯示厚度增加50μm)減少水分流失。利用全景掃描觀察海星再生,記錄斷肢重新發育的細胞分化細節。
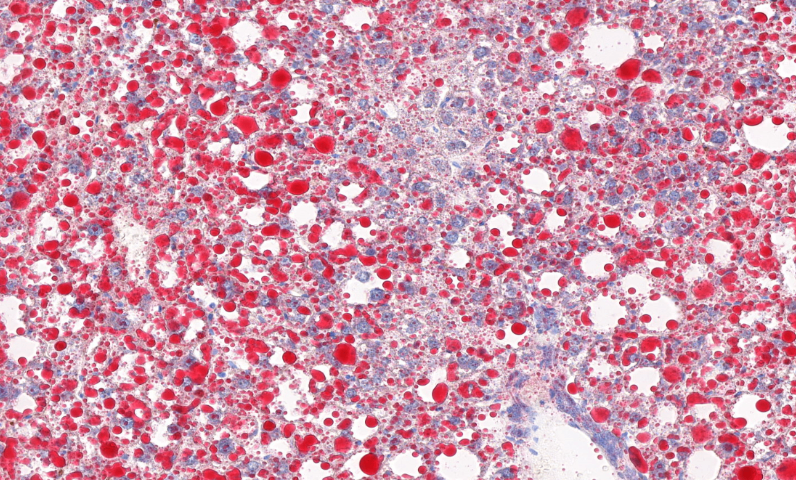
0. 干細胞研究運用全景掃描技術追蹤干細胞的分化潛能與命運決定,通過標記干細胞表面的標志物,實時監測干細胞在不同誘導條件下的分化過程,記錄其向不同細胞類型分化的形態變化及分子表達特征。結合表觀遺傳學分析,揭示干細胞分化的調控機制,例如在胚胎干細胞研究中,全景掃描展示了干細胞在分化為心肌細胞過程中的細胞形態變化及相關基因的表達時序,為干細胞的臨床應用提供了理論基礎,也為再生醫學中細胞替代***提供了細胞來源的制備方法。全景掃描觀察視網膜光適應,記錄感光細胞對光線強度的響應變化。北京腦組織全景掃描
病毒蛋白質組學研究運用全景掃描技術結合蛋白質組學方法。吉林免疫熒光全景掃描
0. 全景掃描在古生物學領域發揮重要作用,借助顯微 CT 與三維重建技術,對化石進行無損傷全景掃描,可清晰呈現化石內部的骨骼結構、牙齒形態甚至軟組織印痕。通過分析這些細節,能推斷古生物的演化關系、生活習性及生存環境,比如對恐龍化石的全景掃描,揭示了不同種類恐龍的骨骼力學特征與運動方式的關聯,為研究恐龍的演化歷程提供了關鍵證據。同時,它還能對比不同地質年代化石的結構變化,追蹤生物演化的關鍵節點,推動對生命起源與演化規律的深入探索。吉林免疫熒光全景掃描
- 浙江雞ELISA試劑盒價格實惠 2025-09-20
- 西藏科研酶聯免疫吸附測定試劑盒大概多少錢 2025-09-20
- 海南熒光全景掃描市場價格 2025-09-20
- 上海進口ELISA抗體試劑怎么樣 2025-09-20
- 河南大鼠科研一抗大概多少錢 2025-09-20
- 湖南兔酶聯免疫吸附測定試劑盒使用方法 2025-09-20
- 中國香港試驗室ELISA試劑盒一般多少錢 2025-09-20
- 湖北試驗室ELISA抗體試劑怎么用 2025-09-20
- 浙江熒光全景掃描銷售電話 2025-09-19
- 福建犬科研一抗電話多少 2025-09-19
- 進口器官芯片protocol 2025-09-20
- 湖北手掌矯形器哪里有 2025-09-20
- 坪山區推薦生物試劑電話多少 2025-09-20
- 長寧區附近教育軟件服務電話 2025-09-20
- 浙江合成文庫技術優勢 2025-09-20
- 虹口區新款醫療管理服務服務費 2025-09-20
- 四川全腸道菌群檢測制劑 2025-09-20
- 松江區包埋產品 2025-09-20
- 福建缺血預適應訓練儀概念 2025-09-20
- 北京推拿訓練儀器生產廠家 2025-09-20